Sequenzierung von SARS-CoV-2 Genomen
Betr. Coronavirus-Surveillanceverordnung
Die am 19.01.2021 in Kraft getretene Coronavirus-Surveillanceverordnung – CoronaSurV verpflichtet nach § 23 Absatz 3 Satz 1 des Infektionsschutzgesetzes Laboratorien und Einrichtungen, die Untersuchungsmaterial und Isolate von Krankheitserregern in Bezug auf das Corona Virus SARS-CoV-2 untersuchen und in diesem Rahmen eine Vollgenomsequenzierung dieses Erregers vornehmen, die erhobenen Sequenzdaten das Robert Koch-Institut zum Zwecke der Krankheitserregersurveillance zu übermitteln.
Für die Übermittlung der Genomsequenzen haben die sequenzierenden Laboratorien und Einrichtungen einen Anspruch auf eine Vergütung in Höhe von 220 € pro Datenübermittlung.
Bei mehr als 70.000 Neuinfektionen innerhalb der jeweils vorausgegangenen Woche können 5% der positiv getesteten Proben sequenziert und eingesendet werden. Bei weniger als 70.000 Neuinfektionen innerhalb der jeweils vorausgegangenen Woche erhöht sich der Anteil auf 10% der positiv getesteten Proben.
Einrichtungen und Laboratorien, die eine SARS-CoV-2 Diagnostik durchführen, jedoch selbst keine Genomsequenzierung vornehmen, können einen bestimmten Anteil der positiv auf SARS-CoV-2 getesteten Proben in andere Laboratorien und Einrichtungen zur Durchführung der Sequenzierung einschicken. In diesem Fall werden Versandkosten erstattet.
Mehr als 10.000 SARS-CoV-2 Genome sequenziert (Januar 2022)
Als Experten für Hochdurchsatzsequenzierung haben wir bereits im April 2020 mehrere Genome des Sars-CoV-2 Virus sequenziert und analysiert (Toptan et al. 2020), und aktuell bereits > 10.000 SARS-CoV-2 Genome sequenziert.
Dank einer gut funktionierenden Probenvorbereitung und Datenanalyse können wir pro Tag bis zu 400 Sars-CoV-2 Genome analysieren. Hierzu benötigen wir lediglich die isolierte RNA positiver Patienten.
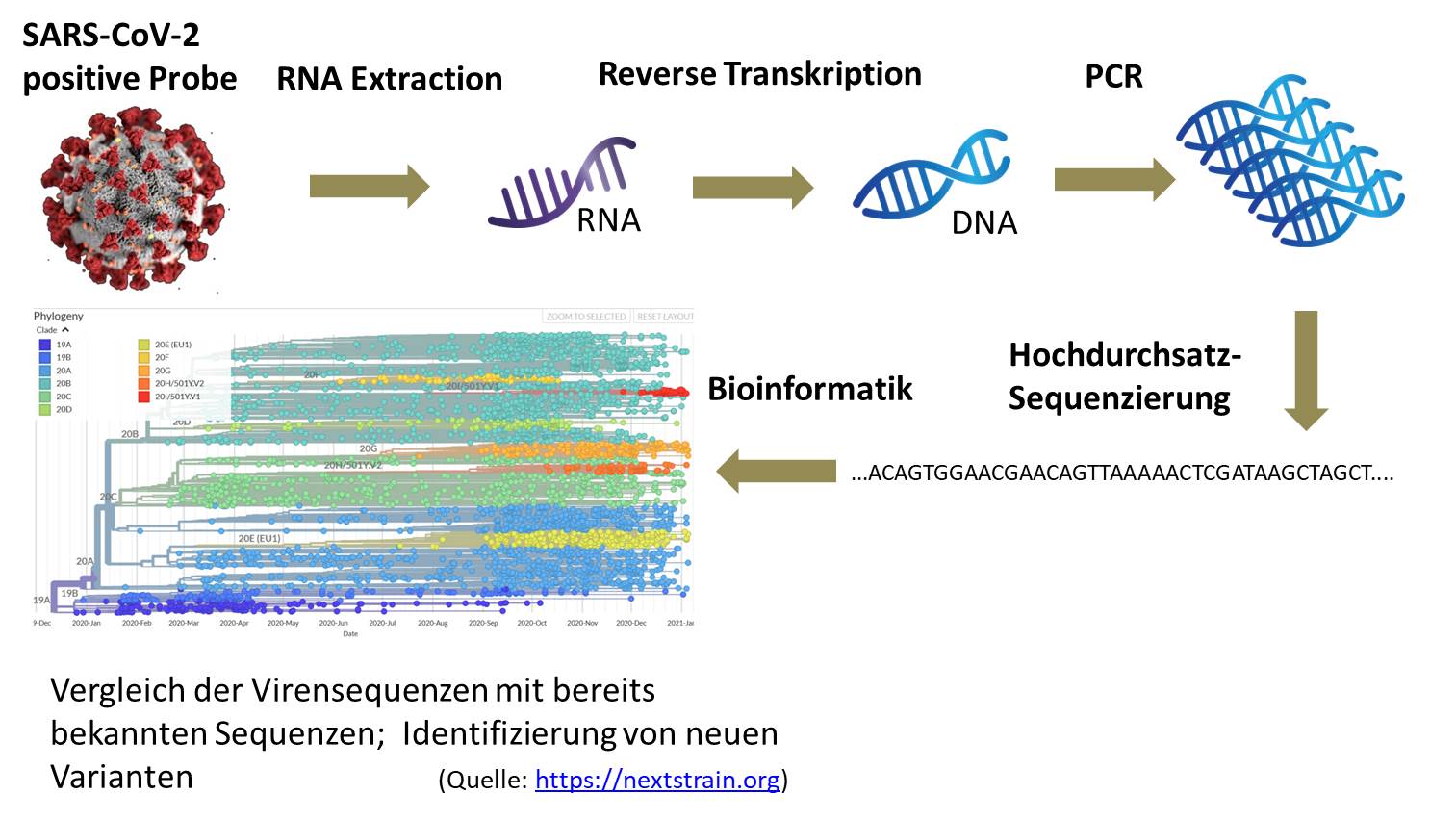
Ablauf der Analyse
Die RNA des Virus von positiv getesteten Patienten wird umgeschrieben in DNA und mittels PCR vermehrt. Hiernach wird die DNA für die Sequenzierung aufbereitet und mittels Hochdurchsatzsequenzierung (“Next Generation Sequencing”) in hoher Abdeckung sequenziert.
Die Daten werden dann dem RKI übermittelt.
Die Sequenzen werden verglichen mit Referenzsequenzen einer Datenbank und Unterschiede werden ermittelt. Bekannte Varianten wie die neue Englische Corona Variante (B.1.1.7) oder die Südafrikansche Variante (B.1.3.5.1 / 501Y.V2) werden erfassst und auch neue Varianten können schnell gefunden werden.
Vorteil unseres Rely Puffers für die SARS-CoV-2 Genomsequenzierung
Die Aufnahme des Virus in unseren Rely Puffer anstelle von Viralem Transfer Medium, PBS oder Wasser, schützt die RNA vor Degradation für viele Wochen – und zusätzlich den Anwender vor einer Infektion, da die Virushülle durch den Puffer zerstört wird. Sollte keine RNA mehr übrig sein nach der qPCR, kann somit auf die Originalprobe zurückgegriffen, und hochwertige RNA gewonnen werden.
Kontakt
Gerne stehen wir für eine Zusammenarbeit mit Diagnoselaboren oder Forschungsinstituten zu Verfügung. Bei Interesse schreiben Sie uns bitte eine E-mail (info@genxpro.de), telefonisch können Sie uns unter der (069) 95739710 erreichen.
Referenz
Optimized qRT-PCR approach for the detection of intra- and extra-cellular SARS-CoV-2 RNAs
Weitere Produkte und Service für SARS-CoV-2:
-
Gleichzeitiger Nachweis von SARS-CoV-2 inklusiver aller Variants of Concern; Quadplex N,M, RP, N501Y qPCR Kit
-
qPCR Kit zum Nachweis von N501Y und del 69-70; Quadplex M,S,N501Y, del69-70, RP
-
qPCR Kit zum Nachweis der E484K Mutation (P1; Südafrika; Supermutante; Impfresistenz; Mehrfachansteckung)
-
Dual Target SARS-CoV-2 qPCR Assay (Englisch)
-
RELY-viral medium: inactivating the virus, stabilizing the RNA at RT (Englisch)
-
Genomseqeunzierung von SARS-CoV-2